- 首页
-
产品中心
仪器设备
试剂耗材
解决方案
-
成像系统
-
核酸提取仪/均质器
-
低温存储生物样本深低温转运桶-CryoPak-Genepoint-基点 半自动液氮存储系统-Hatch Lite-Genepoint-基点 样本库转运机器人-Pelican-Genepoint-基点 全自动液氮存储系统-Hatch-Genepoint-基点 -80℃自动化单体存储系统-Snorkel-Genepoint-基点 -20℃自动化单体存储系统-Snorkel-Genepoint-基点 高效气相液氮罐-Genepoint-基点 智能气相液氮罐-Genepoint-基点 隔氮型液氮罐-V5000EH-AB/C-CBS 隔氮型液氮罐-V3000EH-AB-CBS 隔氮型液氮罐-V1500-AB-CBS 车载便携式超低温冰箱-ULT25NEU-Stirling-斯特林 桌下型超低温冰箱-SU105UE-Stirling-斯特林 大容量超低温冰箱-VAULT100-Stirling-斯特林
-
均相检测HTRF CAMP GS DYNAMIC KIT-Revvity-瑞孚迪 HTRF HUMAN IFN G KIT-Revvity-瑞孚迪 HTRF IP-One Gq Detection Kit-Revvity-瑞孚迪 HTRF ADV.ERK P-T202/Y204 KIT-Revvity-瑞孚迪 HTRF STREPTAVIDIN TB cryptate-Revvity-瑞孚迪 HTRF MAb Anti FLAG M2 XL665-Revvity-瑞孚迪 HTRF MAb Anti GST-Tb cryptate-Revvity-瑞孚迪 HTRF MAB ANTI-6HIS TB GOLD-Revvity-瑞孚迪 HTRF cAMP Gs HiRange Detection Kit-Revvity-瑞孚迪 HTRF STAT3 P-Y705 KIT-Revvity-瑞孚迪 HTRF HUMAN IL2 KIT-Revvity-瑞孚迪 HTRF HUMAN IL6 KIT-Revvity-瑞孚迪 HTRF HUMAN IL8 KIT-Revvity-瑞孚迪 HTRF HUMAN TNF ALPHA KIT-Revvity-瑞孚迪 HTRF KINEASE STK S3 KIT-Revvity-瑞孚迪 HTRF KinEASE-TK Detection Kit-Revvity-瑞孚迪 HTRF Phospho-IR-β (Tyr1150 /1151) Detection Kit-Revvity-瑞孚迪 HTRF Human Phospho-AKT1/2/3 (Ser473) Detection Kit-Revvity-瑞孚迪 HTRF Human Total c-RAF Detection Kit-Revvity-瑞孚迪 HTRF ERK P-T202/Y204 KIT-Revvity-瑞孚迪 HTRF Human and Mouse Total ERK Detection Kit-Revvity-瑞孚迪 HTRF RB P-S807/811 Kit-Revvity-瑞孚迪 AlphaLISA Nickel Chelate Acceptor Beads-Revvity-瑞孚迪 AlphaLISA Anti-DIG Acceptor Beads-Revvity-瑞孚迪 ALPHA.SF ULTRA ERK 1/2 PT202/Y204-Revvity-瑞孚迪 ALPHA.SF ULTRA RAB10 PT73-Revvity-瑞孚迪 ALPHA.SF ULTRA NEW STAT5 PY694/699-Revvity-瑞孚迪 EU-ANTI-P-4E-BP1 (THR37/46)-Revvity-瑞孚迪 LANCE Ultra cAMP Detection Kit-Revvity-瑞孚迪 AlphaScreen GST Detection Kit-Revvity-瑞孚迪 LANCE Eu-W1024 Anti-6His Antibody-Revvity-瑞孚迪
-
细胞因子系列
-
全封闭自动化细胞培养系统耗材
-
间充质干细胞MSC
-
ES/iPSC
-
冻存液
-
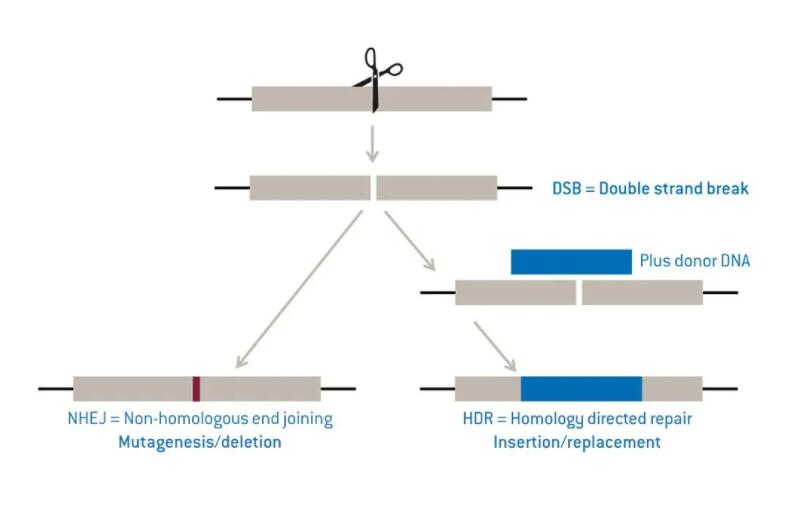
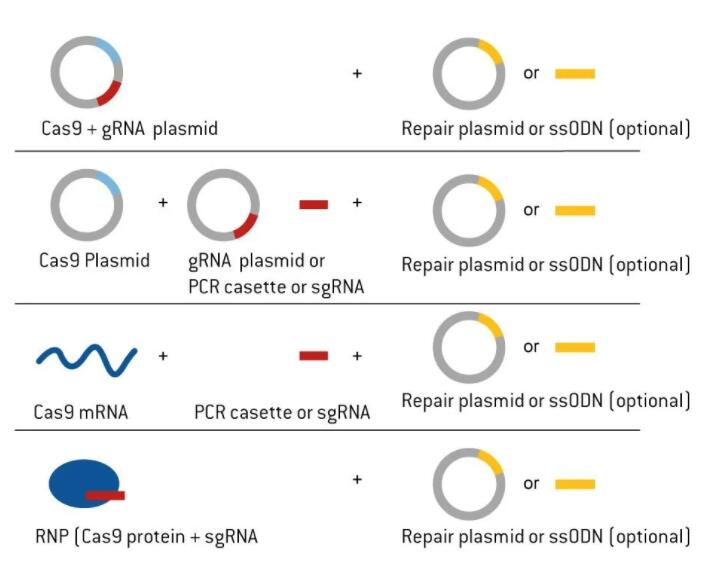
行业解决方案
test
test - 服务支持
- 上海玮驰
- 合作与联系